Validación taxonómica - Species Matching
Objetivo
Realizar una validación taxonómica de nombres científicos de manera automática para detectar posibles errores de formato o taxonomía, usando el servicio Species-matching.
Introducción
Video introductorio de las 3 guías de validación taxonómica, da un contexto general sobre las herramientas y su uso. Si lo prefiere pude realizar la guía siguiendo el paso a paso sin necesidad de ver el video.
Sobre la herramienta
La herramienta Species-matching es un servicio de GBIF que permite validar de manera automática y masiva hasta 5.000 nombres científicos en un conjunto de datos, la herramienta compara los nombres científicos del conjunto contra el árbol taxonómico de GBIF, a partir del cual obtiene la jerarquía taxonómica de cada taxón (kingdom, phylum, class, order, family, genus) y su estatus taxonómico (Sinónimo, Aceptado, Dudoso).
El árbol taxonómico de GBIF está basado en 94 fuentes taxonómicas mundiales, permitiendo la integración de nombres científicos independientemente de la fuente de los datos, registros biológicos, listas o eventos.
Enlace
Species Matching - GBIF: https://www.gbif.org/es/tools/species-lookup
Requerimientos
-
El conjunto de datos debe tener como mínimo una columna llamada
scientificNamecon un nombre científico documentado para todas las filas. -
El conjunto de datos debe estar guardado en formato CSV.
Archivo de trabajo
Descargue el archivo Datos_speciesmatching.zip y descomprimalo para acceder al archivo Datos_speciesmatching.csv con el cual realizará el laboratorio.
Paso 1 - Carga de archivos en Species-Matching
Ingrese a la herramienta en línea Species-Matching y cargue el archivo Datos_speciesmatching.csv (Fig. 1), para hcerlo tiene dos opciones:
- Haga clic en
SELECT FILEy seleccione el archivo desde la ubicación en su ordenador. ó - Arrastre el archivo desde su explorador de archivos y suéltelo en el ícono
DROP HERE.
Para que el archivo sea leído por la herramienta, debe estar en formato .csv y tener como mínimo una columna con nombres científicos llamada
scientificName
Figura 1. Página principal de la herramienta Species Matching.
Paso 2 - Manejo de la herramienta
2.1. Acotar la búsqueda
Luego de cargar el archivo la herramienta le permite al usuario seleccionar un reino (kingdom) contra el cual contrastar los nombres científicos, esto es recomendable cuando todos los registros pertenecen al mismo reino para reducir ambiguedades en la búsqueda.
- Seleccione el reino animalia.
- Haga clic en
MATCH TO GBIF BACKNONE.
Figura 2. Detalle de la selección del reino.
Recomendación: incluya en el conjunto de datos una columna con el elemento
kingdomcuando pertenecen a reinos diferentes y siempre que sea posible en el archivo de validación, de esta forma se evitan problemas ocasionados por homónimos entre distintos reinos ya que la herramienta solo contrastará el nombre científico contra los taxa del reino indicado.
2.2. Resultados de la validación
Una vez validado el archivo, visualizará en la herramienta una tabla con las siguientes columnas/elementos (Fig. 3):
-
verbatimScientificName: el nombre original en el archivo cargado en la herramienta. -
preferedKingdom: reino seleccionado para la validación. -
matchType: tipo de coincidencia entre el nombre científico contra el árbol taxonómico de GBIF. -
confidence: grado de confianza del cruce de información. -
scientificName: nombre científico sugerido de acuerdo al árbol taxonómico de GBIF. -
status: estatus taxonómico del nombre científico. -
rank: rango taxonómico del nombre taxonómico. - Taxonomía superior: Clasificación taxonómica sugerida para:
kingdom,phylum,class,order,familyygenus.
Figura 3. Detalles del resultado de la validación en Species Matching.
2.3. Corrección y edición
Diríjase a la columna matchType, al hacer clic en el encabezado podrá reorganizar los registros de menor a mayor coincidencia (Fig. 4).
Figura 4. Resultados organizados en orden de coincidencia en la columna matchType.
Revise y ajuste los nombres científicos identificados con la etiqueta HIGHERRANK, para ello diríjase a la columna ScientificName, y haga clic en el ícono del lápiz (Fig. 5A), luego aparecerá una ventana que le permitirá seleccionar el nombre científico válido para el registro (Fig. 5B).
Para el registro con nombre Dendrophidium percarinatus, seleccione la segunda opción: Dendrophidion percarinatum (Cope,1893), note que el nombre original tenía un error de tipeo.
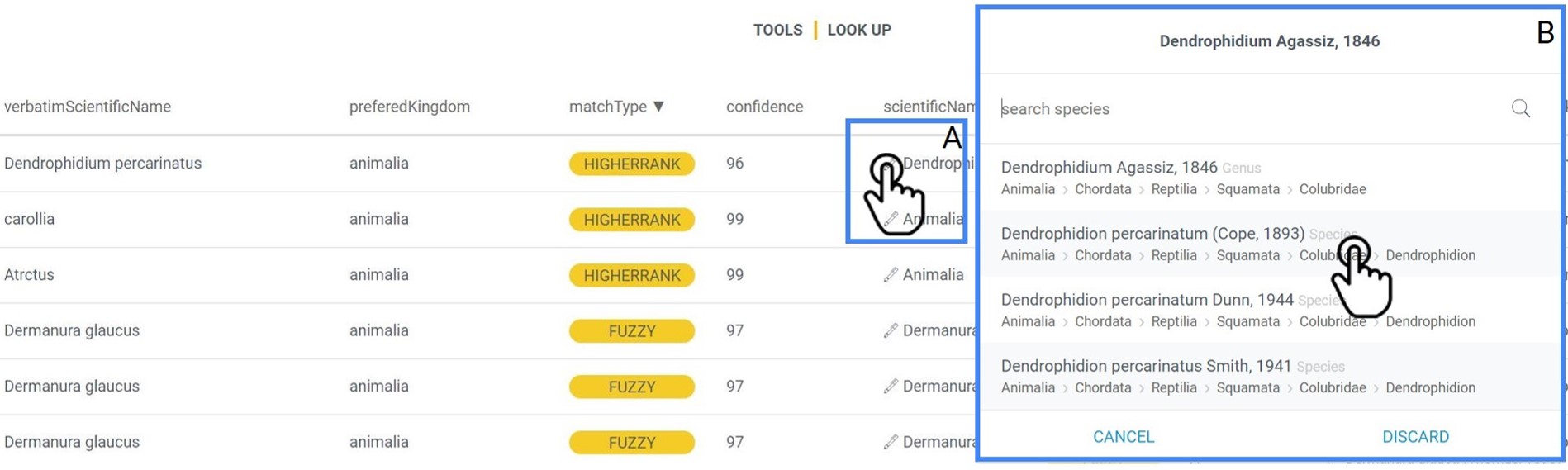
Figura 5. Edición del nombre científico.
La herramienta lápiz le permite editar el nombre interpretado por GBIF para cualquier registro.
No es necesario que ajuste todos los nombres con la etiqueta
FUZZY, esto lo puede realizar de forma masiva en los siguientes pasos.
2.4. Descarga del archivo validado
En la parte inferior derecha encontrará la opción GENERATE CSV (Fig. 6), Haga clic sobre el botón y seleccione la ubicación en su ordenador donde guardará el archivo. El programa descargará un archivo llamado normalized.csv.
Figura 6. Descarga del archivo validado con Species Matching.
Paso 3 - Carga de archivo validado en OpenRefine
En OpenRefine tiene la ventaja de poder realizar filtros y facets que le permitiran explorar con mas detalle los resultados obtenidos de la herramienta Species Matching.
Cargue en OpenRefine el archivo normalized.csv obtenido del paso anterior. Para ello, abra OpenRefine y en el menú lateral y siga la ruta Create Project* > Get data from > This computer > Choose Files. Seleccione el archivo que descargó en el paso anterior y de clic en la opción Next (Fig. 7).
Figura 7. Carga del archivo en Open Refine.
Modifique el nombre del proyecto en la opción Project name dejandolo solamente como normalized. En el campo Character encoding asegurese de seleccionar UTF-8 y finalmente seleccione la opción Create Project(Fig. 8).
Figura 8. Modificación de nombre y creación de proyecto.
Paso 4 - Exploración de resultados en OpenRefine
Explore el archivo que subió a OpenRefine e identifique los elementos matchType, confidence, status y rank, que le permitirán validar y decidir que acciones tomar en caso de que necesite corregir el nombre científico de los registros biológicos (Fig. 9A)
Adicionalmente cuenta con las columnas que contienen la jerarquía taxonómica (Fig. 9B), con las cuales podrá validar y completar la taxonomía superior en su conjunto de datos.
Figura 9. Contenido de las columnas en OpenRefine.
4.1. Creación de filtros en OpenRefine
Realice un Text facet en la columna matchType. Para hacerlo, haga clic en matchType > Facet > Text facet (Fig. 10A). Repita este procedimiento y cree otro Text facet para la columna status.
En el panel lateral izquierdo aparecerá los dos filtros creados (Fig. 10B), explore cuantos registros tiene un nombre científico que coincide de manera exacta EXACT con el árbol taxonómico de GBIF, cuantos coinciden parcialmente FUZZY y cuantos coinciden solo a un nivel taxonómico superior al documentado en el nombre científico HIGHERRANK. Explore también cuantos registros tienen un nombre científico con estatus taxonómico aceptado ACCEPTED y cuántos son sinónimos SYNONYM. Al hacer estos filtros usted podrá identificar aquellos registros cuyo nombre científico tiene errores de tipeo y corregirlos.
Figura 10. Creación de filtros y visualización de resultados en OpenRefine.
4.2. Comparación
Compare los elementos verbatimScientificName (el nombre original en su conjunto de datos) y scientificName (el nombre válido según GBIF) para los registros con matchtype = FUZZY, observará que algunos registros tienen errores de tipeo. Utilice su nuevo conocimiento en OpenRefine para hacer los ajustes que considere pertinentes.
Paso 5 - Datos propios
Si cuenta con un conjunto de datos propio, intente validar los nombres científicos siguiendo los pasos de este laboratorio.
¡Felicitaciones! ![]() Terminó la revisión de los nombres científicos con la herramienta species-matching
Terminó la revisión de los nombres científicos con la herramienta species-matching
Atribución y uso de los laboratorios

La licencia CC-BY te permite usar, redistribuir y construir sobre estos contenidos libremente. ![]() Queremos que compartas estos laboratorios y que juntos logremos datos sobre biodiversidad de mejor calidad.
Queremos que compartas estos laboratorios y que juntos logremos datos sobre biodiversidad de mejor calidad.
Citación sugerida
Plata C., Ortíz R., Marentes E. (2021). Laboratorio de datos, Ciclo de formación. Consultado a través del SiB Colombia. Disponible en https://sib-colombia.github.io/Formacion/